Les techniques de séquençages haut-débit ou HTS
Thématiques
Les techniques de séquençages haut-débit ou HTS (High Throughput Sequencing) se sont développées au travers du projet ‘Génome humain’ lancé en 1988. Depuis les HTS se sont très largement diffusées dans tous les domaines de la biologie.
Les HTS ont la capacité théorique de détecter tout acide nucléique (information génétique) au sein d’un échantillon, dépendant de la méthode d’extraction et de purification choisie. Les coûts de séquençage HTS ont drastiquement diminué permettant leur appropriation et leur utilisation en santé des plantes.
La virologie de la vigne intègre depuis une dizaine d’années ces méthodologies dans ses questions de recherche mais encore très peu dans les domaines de la certification ou de la ‘qualité sanitaire’. Ceci est très important sachant que la vigne héberge un grand nombre de virus et en général de façon simultanée, et qui constitue son virome.
Seuls quelques virus parmi eux sont connus pour causer ou être associés à des syndromes graves sur vigne, et font d’ailleurs partie du schéma de certification. Cependant, pour la très grande majorité des virus appartenant à ce virome, leur impact sur la vigne est peu, voir pas connu.
On peut donc de façon légitime se poser les questions suivantes : Faut-il tous les identifier ? Pourquoi ? Faut-il éliminer tous les virus ? Certains peuvent-ils être bénéfiques ? Peut-on utiliser les HTS pour anticiper des émergences virales ? Une réponse possible peut être apportée considérant les émergences virales comme ‘danger potentiel’ (à l’instar de la problématique lié au SARS-CoV-2 et la Covid-19 chez l’être humain).
Bien que révolutionnaire dans leur approche, ces techniques HTS ne sont pas encore complètement optimisées sur vigne et des travaux de R&D doivent être poursuivis afin de lever certains verrous techniques.
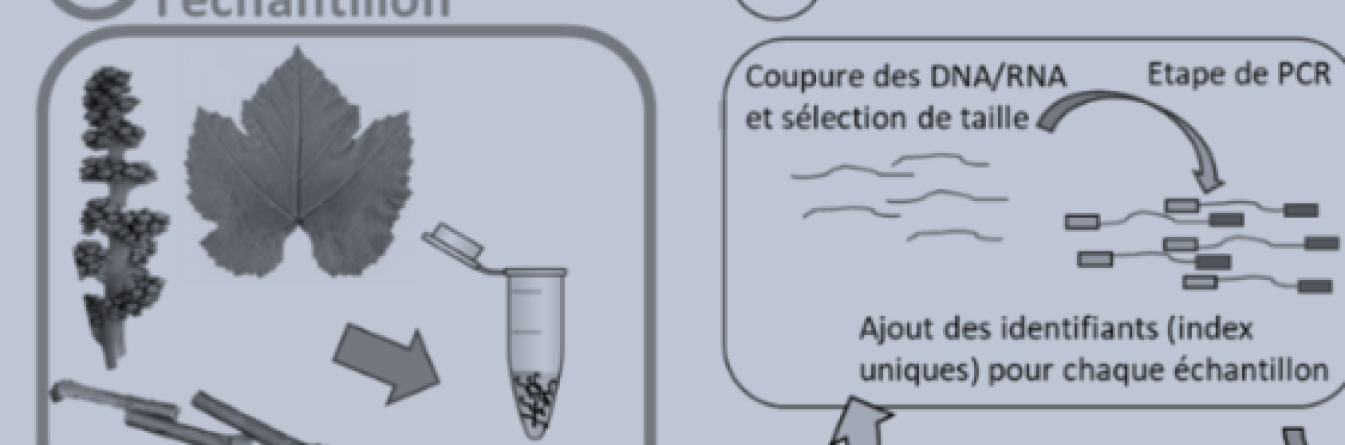
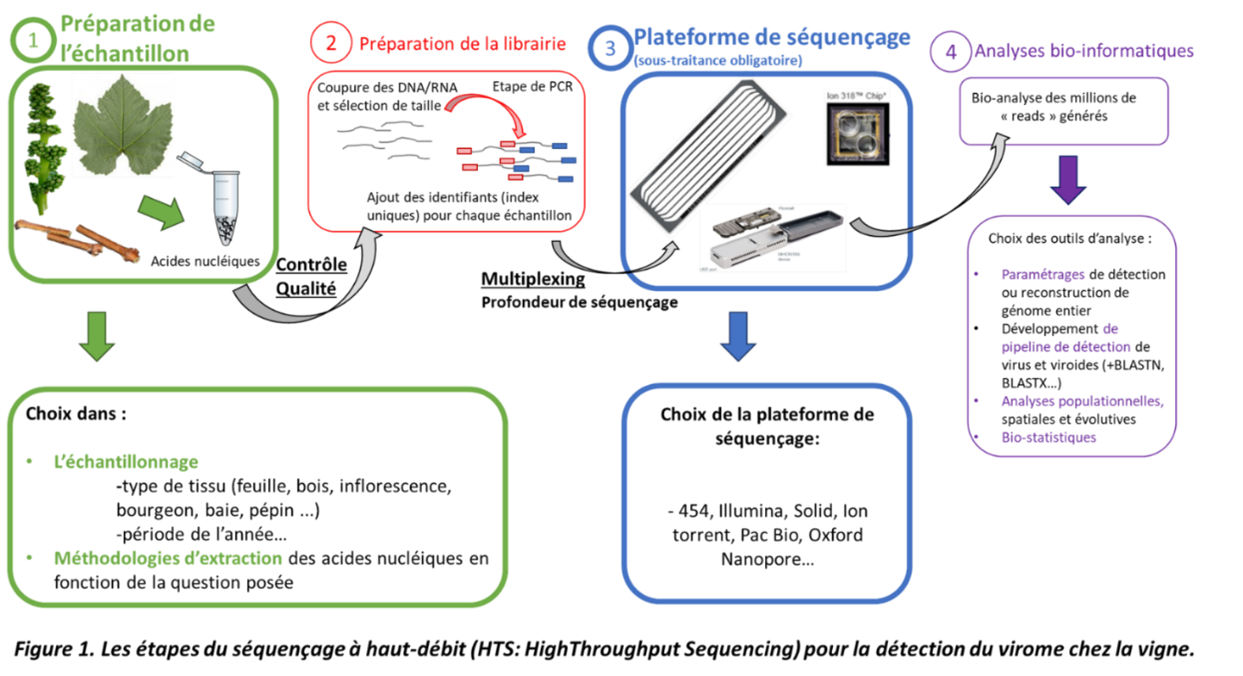
Les analyses HTS peuvent être scindées en quatre grandes étapes successives, de la préparation des échantillons à l’obtention des résultats après analyses bio-informatiques (Figure 1, étapes de 1 à 4). Chacune de ces étapes peut et doit être optimisée dans un objectif d’utilisation en routine.
Le projet OptiSeq mené en collaboration entre l’IFV et l’INRAE et financé par le CASDAR, se propose d’étudier et d’optimiser plus spécifiquement les étapes du processus concernant la préparation de l’échantillon avant le séquençage en lui-même (amélioration des protocoles d’échantillonnage, d’enrichissement de l’information génétique ainsi que sur le choix de la technique de séquençage, étapes 1 et 3 dans la Figure 1).
L’optimisation des pipelines d’analyse (étape 4) fera l’objet d’un autre projet en cours d’évaluation.
Contact : Jean-Michel Hily (jean-michel.hily@vignevin.com), Olivier Lemaire (olivier.lemaire@inrae.fr).
Vous pourriez être intéressé par ...
- Article webzineAjouté le 03/09/2018
